Covid-19, supercomputer individua molecole che inibiscono l'ingresso del virus nelle cellule
La ricerca del Gruppo di Bioinformatica di "Tor Vergata" su "Virus Research"
Uno studio italiano, realizzato interamente dal gruppo di ricerca in Bioinformatica strutturale del Dipartimento di Biologia dell’Università Roma “Tor Vergata” e pubblicato online per la rivista internazionale “Virus Research” (Elsevier), ha preso in esame la cavità situata nella parte inferiore della proteina di membrana del virus SARS-CoV-2, la macromolecola Spike S glycoprotein, invece della parte superiore come proposto da molti.
L’articolo “Targeting the SARS-CoV-2 spike glycoprotein prefusion conformation: virtual screening and molecular dynamics simulations applied to the identification of potential fusion inhibitors” (https://doi.org/10.1016/j.virusres.2020.198068) è disponibile sulla piattaforma “ScienceDirect” e su Pubmed. Si tratta del primo lavoro interamente computazionale sottoposto a revisione tra pari che prende in considerazione come bersaglio per l’inibizione una porzione interna di Spike S glycoprotein, glicoproteina di membrana del virus SARS-CoV-2 che, a seguito del legame con il recettore ACE2 della cellula umana, dà inizio al processo infettivo.
Lo scopo del lavoro è stato quello di trovare, utilizzando delle tecniche computazionali allo stato dell’arte, delle molecole appartenenti al gruppo delle “FDA approved drugs”, ovvero farmaci approvati dalla Food and Drugs Administration Americana e dunque facilmente riutilizzabili perché già noti, avvalendosi della procedura cosiddetta di “drug repurposing”. Tali farmaci dovrebbero essere capaci di bloccare la proteina Spike mentre esegue la “transizione conformazionale”, ovvero il movimento molecolare necessario al completamento del processo infettivo.
Ma bloccarla dove? «Le molecole inibitrici - ci spiega il prof. Mattia Falconi, Coordinatore della Laurea Magistrale in Bioinformatica, tra gli autori della ricerca - sfruttano la cavità interna della glicoproteina Spike e occludendola impediscono il movimento dell’intera macromolecola».
Gli studi realizzati dal Gruppo di ricerca di Bioinformatica Strutturale di “Tor Vergata, coordinato dal professor Falconi, si sono concentrati sulla cavità interna del recettore Spike. «Attraverso uno screening virtuale abbiamo osservato che alcuni farmaci possono occludere questa cavità impedendo così il movimento dell’intera macromolecola e bloccando il processo infettivo messo in atto dalla glicoproteinadel virus SARS-CoV-2».
«L’aspetto molto interessante - continua Falconi - è che la zona utilizzata come bersaglio dagli inibitori rimane sostanzialmente invariata nelle sequenze genomiche conosciute di SARS-CoV-2 a oggi disponibili, e possiede un alto grado di similarità anche con quelle di altri betacoronavirus, in quanto non sottoposta a fenomeni di mutazione. Infatti, un’alterazione di questa regione impedirebbe la fase iniziale del meccanismo di infezione e porterebbe alla scomparsa del virus. L’efficacia di queste molecole inibitrici sarà testata a breve attraverso tecniche sperimentali in laboratorio, su pseudo-virus».
Lo screening virtuale è stato eseguito grazie all’utilizzo del cluster HPC dell'ENEA CRESCO6, il supercomputer messo a disposizione da ENEA per la ricerca su Covid-19.
In questo studio è stato valutato per la prima volta, a livello computazionale, un promettente approccio terapeutico atto a prevenire la diffusione del virus SARS-CoV-2 e basato sulla ricerca di composti in grado di inibire specificatamente i cambiamenti conformazionali innescati nella glicoproteina di membrana del virus (Spike S) a seguito del suo legame con il recettore cellulare ACE2. Dopo questo legame, infatti, due regioni chiave della macromolecola Spike, chiamate HR1 e HR2 (presenti in triplice copia, data la struttura trimerica della proteina), subiscono profondi cambiamenti conformazionali, assumendo una forma simile a un fuso che permette l’associazione del virus con la cellula ospite (vedi Fig. 1).
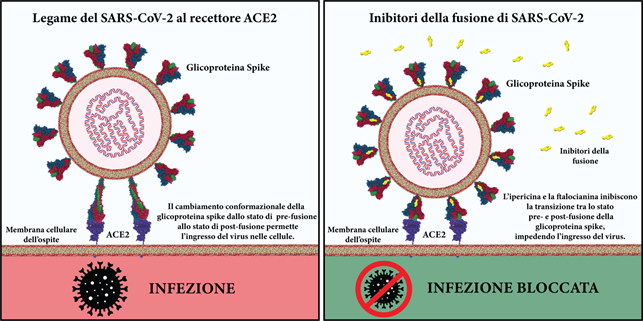
Fig. 1 - A sinistra il virus SARS CoV-2 in assenza dei farmaci proposti come inibitori del meccanismo di infezione; a destra in presenza di molecole selezionate dal virtual screening (rappresentate in giallo) che si legano alla glicoproteina del virus. A sinistra quando le Spike contattano ACE2 cambiano forma e diventano affusolate (subiscono una transizione conformazionale), dando il via al processo di infezione. Invece, a destra, quando le Spike contattano ACE2 della cellula umana in presenza del farmaco, rimangono nella stessa forma globulare e il processo infettivo si arresta.
«L’obiettivo del nostro studio - affermano la dott.ssa Alice Romeo e il dott. Federico Iacovelli, PhD,ricercatori delDipartimento di Biologia di “Tor Vergata” ed entrambi primi coautori della ricerca, è stato identificare possibili composti che possano legarsi stabilmente nella cavità interna della Spike S di SARS-CoV-2 e agire come inibitori della fusione».
I composti per i quali sono state ottenute le migliori energie di legame sono la ftalocianina e l’ipericina, entrambi precedentemente valutati in diversi studi come possibili antivirali su diversi tipi di virus, tra cui l’HIV. «Data la struttura trimerica della Spike, abbiamo ipotizzato che almeno tre molecole possano legarsi contemporaneamente all’interno della cavità, a livello di ognuna delle tre regioni HR1 interne, creando una forte rete di interazioni e un ingombro sterico tale da portarci a ipotizzare che la loro presenza possa indurre la stabilizzazione delle regioni interne contattate, impedendone i rimodellamenti conformazionali necessari alla Spike per la transizione alla conformazione di postfusione».
«Attraverso allineamenti di sequenza tra la glicoproteina Spike utilizzata nel nostro studio e tutte le sequenze genomiche di SARS-CoV-2 e di altri betacoronavirus depositate nel database GenBank (https://www.ncbi.nlm.nih.gov/genbank/) è stato evidenziato che la sequenza della regione HR1 interna alla cavità, target dei nostri composti, è completamente conservata tra diversi isolati, ovvero genomi sequenziati in momenti e luoghi diversi nel mondo, di SARS-CoV-2 e anche tra diversi isolati di SARS-CoV e di altri virus animali simili. I risultati ottenuti possono quindi essere estesi a tutti gli isolati di SARS-CoV-2 a oggi sequenziati e potrebbero avere un’applicazione anche nel trattamento di altri tipi di infezioni virali», concludono i ricercatori Romeo e Iacovelli.
Per maggiori info leggi l’articolo pubblicato su:
- PubMeb https://pubmed.ncbi.nlm.nih.gov/32565126/
- ScienceDirect https://www.sciencedirect.com/science/article/pii/S016817022030383X
A cura dell'Ufficio Stampa di Ateneo

